本文在“自然通信”期刊上发表。由于染色质,DNA复合物和蛋白质,将近两米的DNA线置于人体细胞的小核中,使其变为紧凑,但复杂的结构。为了研究包装DNA的方法,通过全世界的科学家通过所谓的染色体(3C)的构象来使用,并且它们的最具生产力之一是Hi-C方法。它允许您使用高性能测序检测整个基因组的DNA的触点。
然而,在此,存在问题:Hi-C工作需要数十种DNA微克 - 即,具有独特的空间染色质组织的数百万个细胞。此信息必须平均才能获得不考虑单个细胞中DNA包装的特征的常见图片。
正如“普通人”实际上没有存在,传统的HI-C方法不能显示,在同一细胞中同时发生DNA部分的多种相互作用。此外,这种“集体肖像”几乎没有帮助了解物理过程导致形成染色质三维结构的形成。
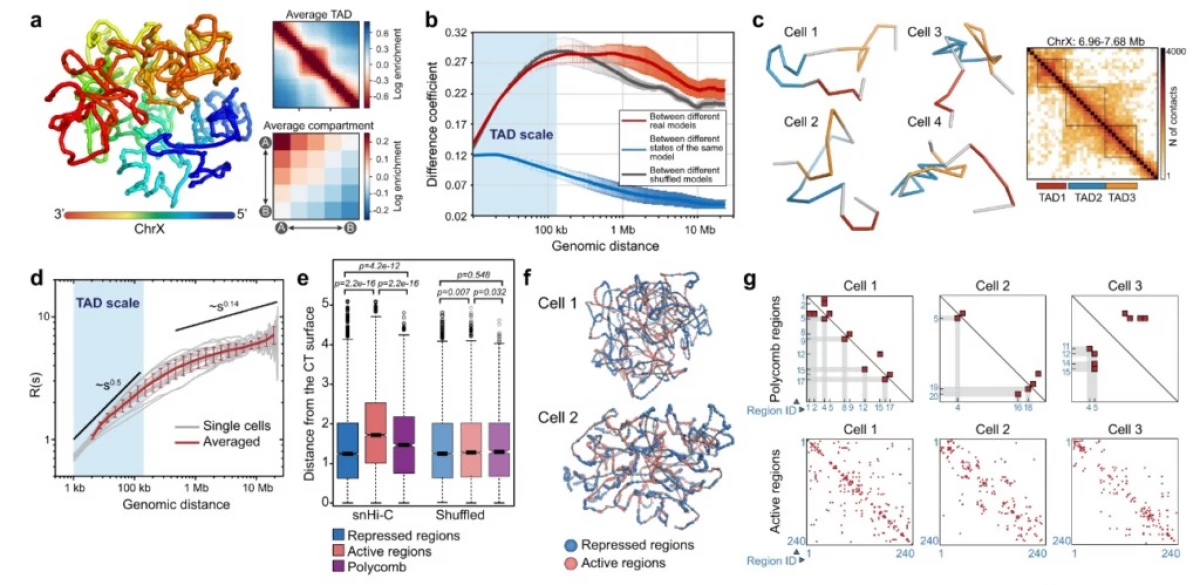
“我们看到一些结构,例如所谓的拓扑相关域(塔德达),在平均DNA接触卡中,但我们不知道它们是否存在于个体细胞中,或者这些是伪像平均值。此外,我们知道,从基因表达的角度来看,即使在同一组织的细胞中也发生了各种各样的 - 来自这里的自然问题是关于它们在结构层中多样化的自然问题,“共同根据生物医学研究,Mikhail Gelfand的作者副总裁斯科拉纳。
为了解决这些问题并使实验HI-C更适合个体细胞,若干机构的研究人员开发了一种称为Hi-C单细胞的方法。由Gelfand和Life Scoop Charch中心副教授领导的Skoltech团队凯瑟琳教堂提供了一项任务,以优化Hi-C单细胞的数据处理,并探索果蝇细胞的基本性质。
他们的同事来自俄罗斯科院和莫斯科国立大学的生物学研究所,以MV LOMONOSOV命名,与俄国法国跨学科科学中心PONCELE的员工一起进行了优化的方法,使其适用于果蝇细胞的实验。
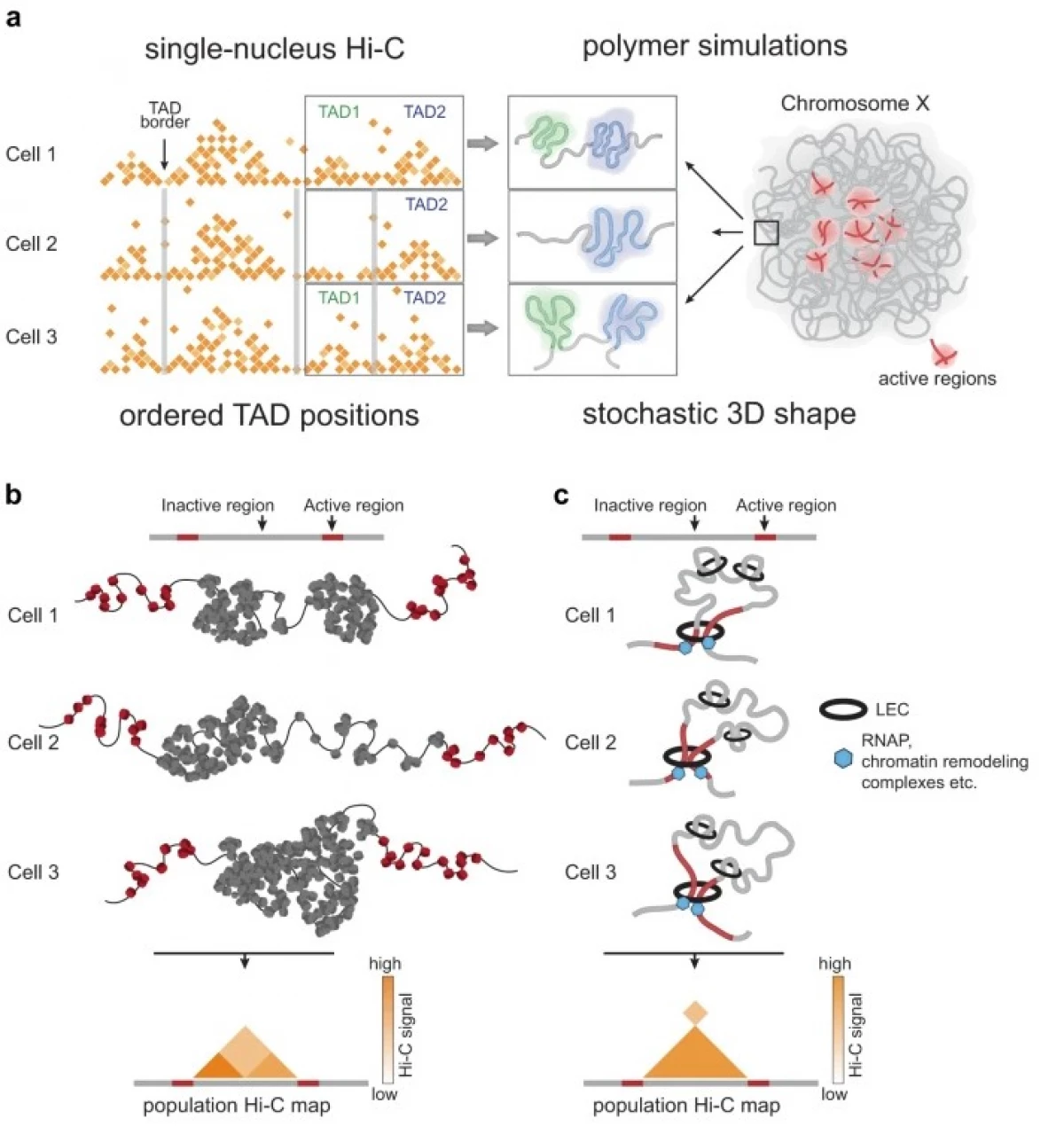
该团队开始采用HI-C方法的标准步骤,其中染色质的结构是化学固定的,DNA被切割并“重新组装”,使得在自然条件下的碎片靠近,结果是“缝制“。但随后而不是一次使用所有DNA,科学家使用PHI29噬菌体聚合酶从每个细胞中扩增微量DNA。该聚合酶通常在扩增DNA时使用,部分地,由于其甚至在非常小的样品上产生大量DNA,而不是比其他普遍的聚合酶更小的误差。
然而,它证明,这种方便的DNA聚合酶尽管复制的精度相当高,可以在DNA分子之间“跳跃”,从而产生人工键,即Hi-C算法不能与真实的相互作用区分。因此,研究人员必须提出一种抑制聚合酶随机“跳跃”的机制。
它们在果蝇细胞上使用了新方法,以了解各种生物是否具有染色质包装的共同基本原理。以前关于哺乳动物细胞的研究表明,仅在群体Hi-C获得的接触卡上存在TADE,但不存在于单个细胞中。然而,果蝇细胞的研究表明,这些结构域在每个特定细胞中。
要了解哪种生物机制负责形成这些可持续域,将需要额外的研究;虽然科学家提供了两种型号的发生。其中一个表明,通过“粘性”的机制组织了果蝇中的染色质,即其部分部分更可能彼此连接。根据另一个,描述所谓的铰链挤出机制,大蛋白质复合物从DNA螺纹产生环,并且由于该封装DNA。
“也许最有趣的问题之一是折叠染色质规则在不同类型的生物体中是相同的。使用单细胞的Hi-C方法,Droozobils,我们发现类似于哺乳动物细胞中的结构域的结构域也存在于该昆虫的基因组中。然而,这些结构比哺乳动物更令人有序,“亚历山大大学,Scholtech研究生和第一个作者之一。
“我们将继续研究染色质的结构和形成环和Tadov的机制。在没有答案的情况下,这个领域还有很多问题。我们已经知道,某些生物体中的这些机制可能会有所不同,但染色质折叠整体的进化是什么?如果我们想要以足够的细节理解这一点,我们需要填补空白,研究染色体在奇怪的生物中的结构,而不仅仅是那些已经很好地调查的差距。因此,我们已经使用了海滨海绵,酵母和amoebas,“Catherine Temmeva说。
根据她,该集团也从事染色体组织的变化与疾病,身体和老化的发展可能的联系。 “如果我们认为染色体的建筑与基因的表达密切相关,那么,响应这些问题,我们将能够处理人体发展,衰老和疾病的制定,”德国说。
俄罗斯州科学院校Gena的生物学研究所专家,莫斯科国立大学以M.V. Lomonosov,法国科学研究中心,俄国法国跨学科科学中心PONCELE等组织参与其中。
来源:裸体科学
